
临时分享测试用,请在复制粘贴后将后缀改为
%{
Method Tasks : 将输入的DNA序列翻译为氨基酸序列
-----------------------------------------------------------------------
Parameters : seq,kind,num,base_number,long_peptide,flag
ReturnValues : peptide
Called By : none
Calls : none
Author : 叶边月、[email protected]
Date : from 2020-9-16
Last update time: 2020-9-20
Version: 3.0
-----------------------------------------------------------------------
TODO:
1.对基因序列中的每个碱基的数目进行可视化
2.增加启动子识别机制
3.生成另一条链上的三条链
4.精简代码,通过函数简化重复操作,通过哈希值的方法进行启动子翻译的过程
5.封装为软件or部署至网站,美化UI,使观感舒适
DONE:
1.对基因序列中每个碱基的数目进行统计
2.统一输入DNA序列字母大小写
3.识别终止子(具体为如何通过break跳出多层嵌套)
4.针对不同的*三种解释方法*进行分类
5.修复seq函数会在一定情况下出现数组元素溢出的情况
Problem still exist:
1.flag=1部分可执行,但无法输出通知信息
-----------------------------------------------------------------------
History :
1.0:2020.9.18:碱基统计机制完成。
2.0:2020.9.19:启动子翻译机制完成基本流程,统一输入DNA序列字母大小写。
3.0:2020.9.20:完成翻译机制三种翻译方式的部分,对该源码进行注释。
%}
%_______________________________________________________________________
%定义变量
kind=["A","T","C","G"];
%输入部分
seq=upper(input("Please input the DNA sequence: ",'s'))
%定义函数&处理部分
%%1.密码子对应
%%%暂时弃用,直接在后续过程中进行判断操作,后续将通过自建函数进行优化。
%%2.统计碱基数量&输出统计结果
for i=1:length(kind)
num(i)=length(strfind(seq,kind(i)));
end
fprintf("The number of A is %d\n",num(1))
fprintf("The number of T is %d\n",num(2))
fprintf("The number of C is %d\n",num(3))
fprintf("The number of G is %d\n",num(4))
base_number=num(1)+num(2)+num(3)+num(4);
fprintf("The Number of bases in DNA is %d\n",base_number)
%%3.三种翻译方式
%%%%定义参数
%%%%第一种解释方式
long_peptide = (base_number-mod(base_number,3))/3; %计算肽链长度,为定义空白字符串数组做准备
fprintf("#1.The length of peptide ignore Terminator is %d\n",long_peptide)%输出肽链长度信息
peptide=" ";%生成空白字符串数组,以便将对应的字符附加于其上
for i=1:long_peptide %翻译操作
flag=0; %与后面的"flag=1"对应,实现终止子功能
if seq(3*i-2)=="A"
if seq(3*i-1)=="A"
if seq(3*i)=="A"
peptide=sprintf('%s%s',peptide,"K");
elseif seq(3*i)=="T"
peptide=sprintf('%s%s',peptide,"N");
elseif seq(3*i)=="C"
peptide=sprintf('%s%s',peptide,"N");
elseif seq(3*i)=="G"
peptide=sprintf('%s%s',peptide,"K");
end
end
if seq(3*i-1)=="T"
if seq(3*i)=="A"
peptide=sprintf('%s%s',peptide,"I");
elseif seq(3*i)=="T"
peptide=sprintf('%s%s',peptide,"I");
elseif seq(3*i)=="C"
peptide=sprintf('%s%s',peptide,"I");
elseif seq(3*i)=="G"
peptide=sprintf('%s%s',peptide,"M");
end
end
if seq(3*i-1)=="C"
if seq(3*i)=="A"
peptide=sprintf('%s%s',peptide,"T");
elseif seq(3*i)=="T"
peptide=sprintf('%s%s',peptide,"T");
elseif seq(3*i)=="C"
peptide=sprintf('%s%s',peptide,"T");
elseif seq(3*i)=="G"
peptide=sprintf('%s%s',peptide,"T");
end
end
if seq(3*i-1)=="G"
if seq(3*i)=="A"
peptide=sprintf('%s%s',peptide,"R");
elseif seq(3*i)=="T"
peptide=sprintf('%s%s',peptide,"S");
elseif seq(3*i)=="C"
peptide=sprintf('%s%s',peptide,"S");
elseif seq(3*i)=="G"
peptide=sprintf('%s%s',peptide,"R");
end
end
end
if seq(3*i-2)=="T"
if seq(3*i-1)=="A"
if seq(3*i)=="A"
flag=1;
break;
elseif seq(3*i)=="T"
peptide=sprintf('%s%s',peptide,"Y");
elseif seq(3*i)=="C"
peptide=sprintf('%s%s',peptide,"Y");
elseif seq(3*i)=="G"
flag=1;
break;
end
end
if seq(3*i-1)=="T"
if seq(3*i)=="A"
peptide=sprintf('%s%s',peptide,"L");
elseif seq(3*i)=="T"
peptide=sprintf('%s%s',peptide,"F");
elseif seq(3*i)=="C"
peptide=sprintf('%s%s',peptide,"F");
elseif seq(3*i)=="G"
peptide=sprintf('%s%s',peptide,"L");
end
end
if seq(3*i-1)=="C"
if seq(3*i)=="A"
peptide=sprintf('%s%s',peptide,"S");
elseif seq(3*i)=="T"
peptide=sprintf('%s%s',peptide,"S");
elseif seq(3*i)=="C"
peptide=sprintf('%s%s',peptide,"S");
elseif seq(3*i)=="G"
peptide=sprintf('%s%s',peptide,"S");
end
end
if seq(3*i-1)=="G"
if seq(3*i)=="A"
flag=1;
break;
elseif seq(3*i)=="T"
peptide=sprintf('%s%s',peptide,"C");
elseif seq(3*i)=="C"
peptide=sprintf('%s%s',peptide,"C");
elseif seq(3*i)=="G"
peptide=sprintf('%s%s',peptide,"W");
end
end
end
if seq(3*i-2)=="C"
if seq(3*i-1)=="A"
if seq(3*i)=="A"
peptide=sprintf('%s%s',peptide,"Q");
elseif seq(3*i)=="T"
peptide=sprintf('%s%s',peptide,"H");
elseif seq(3*i)=="C"
peptide=sprintf('%s%s',peptide,"H");
elseif seq(3*i)=="G"
peptide=sprintf('%s%s',peptide,"Q");
end
end
if seq(3*i-1)=="T"
if seq(3*i)=="A"
peptide=sprintf('%s%s',peptide,"L");
elseif seq(3*i)=="T"
peptide=sprintf('%s%s',peptide,"L");
elseif seq(3*i)=="C"
peptide=sprintf('%s%s',peptide,"L");
elseif seq(3*i)=="G"
peptide=sprintf('%s%s',peptide,"L");
end
end
if seq(3*i-1)=="C"
if seq(3*i)=="A"
peptide=sprintf('%s%s',peptide,"P");
elseif seq(3*i)=="T"
peptide=sprintf('%s%s',peptide,"P");
elseif seq(3*i)=="C"
peptide=sprintf('%s%s',peptide,"P");
elseif seq(3*i)=="G"
peptide=sprintf('%s%s',peptide,"P");
end
end
if seq(3*i-1)=="G"
if seq(3*i)=="A"
peptide=sprintf('%s%s',peptide,"R");
elseif seq(3*i)=="T"
peptide=sprintf('%s%s',peptide,"R");
elseif seq(3*i)=="C"
peptide=sprintf('%s%s',peptide,"R");
elseif seq(3*i)=="G"
peptide=sprintf('%s%s',peptide,"R");
end
end
end
if seq(3*i-2)=="G"
if seq(3*i-1)=="A"
if seq(3*i)=="A"
peptide=sprintf('%s%s',peptide,"E");
elseif seq(3*i)=="T"
peptide=sprintf('%s%s',peptide,"D");
elseif seq(3*i)=="C"
peptide=sprintf('%s%s',peptide,"D");
elseif seq(3*i)=="G"
peptide=sprintf('%s%s',peptide,"E");
end
end
if seq(3*i-1)=="T"
if seq(3*i)=="A"
peptide=sprintf('%s%s',peptide,"V");
elseif seq(3*i)=="T"
peptide=sprintf('%s%s',peptide,"V");
elseif seq(3*i)=="C"
peptide=sprintf('%s%s',peptide,"V");
elseif seq(3*i)=="G"
peptide=sprintf('%s%s',peptide,"V");
end
end
if seq(3*i-1)=="C"
if seq(3*i)=="A"
peptide=sprintf('%s%s',peptide,"A");
elseif seq(3*i)=="T"
peptide=sprintf('%s%s',peptide,"A");
elseif seq(3*i)=="C"
peptide=sprintf('%s%s',peptide,"A");
elseif seq(3*i)=="G"
peptide=sprintf('%s%s',peptide,"A");
end
end
if seq(3*i-1)=="G"
if seq(3*i)=="A"
peptide=sprintf('%s%s',peptide,"G");
elseif seq(3*i)=="T"
peptide=sprintf('%s%s',peptide,"G");
elseif seq(3*i)=="C"
peptide=sprintf('%s%s',peptide,"G");
elseif seq(3*i)=="G"
peptide=sprintf('%s%s',peptide,"G");
end
end
end
if flag==1;
fprintf("Be careful,met the terminator when the Ordinal is %d",i)
break;
end
end
peptide=strrep(peptide," ","");%去除空白字符串数组生成时的空格
fprintf("The first type for sequence of peptide is %s\n",peptide) %输出肽链氨基酸信息
%%%%第二种解释方式
long_peptide = (base_number-1-mod(base_number-1,3))/3; %计算肽链长度,为定义空白字符串数组做准备
fprintf("#2.The length of peptide ignore Terminator is %d\n",long_peptide)%输出肽链长度信息
peptide=" ";%生成空白字符串数组,以便将对应的字符附加于其上
for i=1:base_number-1%通过去掉基因序列的第一个字符模拟密码子不读取第一个碱基的情况
seq(i)=seq(i+1);
end
seq(i+1)="";%去掉字符串数组中多余的最后一个字符
for i=1:long_peptide %翻译操作
flag=0; %与后面的"flag=1"对应,实现终止子功能
if seq(3*i-2)=="A"
if seq(3*i-1)=="A"
if seq(3*i)=="A"
peptide=sprintf('%s%s',peptide,"K");
elseif seq(3*i)=="T"
peptide=sprintf('%s%s',peptide,"N");
elseif seq(3*i)=="C"
peptide=sprintf('%s%s',peptide,"N");
elseif seq(3*i)=="G"
peptide=sprintf('%s%s',peptide,"K");
end
end
if seq(3*i-1)=="T"
if seq(3*i)=="A"
peptide=sprintf('%s%s',peptide,"I");
elseif seq(3*i)=="T"
peptide=sprintf('%s%s',peptide,"I");
elseif seq(3*i)=="C"
peptide=sprintf('%s%s',peptide,"I");
elseif seq(3*i)=="G"
peptide=sprintf('%s%s',peptide,"M");
end
end
if seq(3*i-1)=="C"
if seq(3*i)=="A"
peptide=sprintf('%s%s',peptide,"T");
elseif seq(3*i)=="T"
peptide=sprintf('%s%s',peptide,"T");
elseif seq(3*i)=="C"
peptide=sprintf('%s%s',peptide,"T");
elseif seq(3*i)=="G"
peptide=sprintf('%s%s',peptide,"T");
end
end
if seq(3*i-1)=="G"
if seq(3*i)=="A"
peptide=sprintf('%s%s',peptide,"R");
elseif seq(3*i)=="T"
peptide=sprintf('%s%s',peptide,"S");
elseif seq(3*i)=="C"
peptide=sprintf('%s%s',peptide,"S");
elseif seq(3*i)=="G"
peptide=sprintf('%s%s',peptide,"R");
end
end
end
if seq(3*i-2)=="T"
if seq(3*i-1)=="A"
if seq(3*i)=="A"
flag=1;
break;
elseif seq(3*i)=="T"
peptide=sprintf('%s%s',peptide,"Y");
elseif seq(3*i)=="C"
peptide=sprintf('%s%s',peptide,"Y");
elseif seq(3*i)=="G"
flag=1;
break;
end
end
if seq(3*i-1)=="T"
if seq(3*i)=="A"
peptide=sprintf('%s%s',peptide,"L");
elseif seq(3*i)=="T"
peptide=sprintf('%s%s',peptide,"F");
elseif seq(3*i)=="C"
peptide=sprintf('%s%s',peptide,"F");
elseif seq(3*i)=="G"
peptide=sprintf('%s%s',peptide,"L");
end
end
if seq(3*i-1)=="C"
if seq(3*i)=="A"
peptide=sprintf('%s%s',peptide,"S");
elseif seq(3*i)=="T"
peptide=sprintf('%s%s',peptide,"S");
elseif seq(3*i)=="C"
peptide=sprintf('%s%s',peptide,"S");
elseif seq(3*i)=="G"
peptide=sprintf('%s%s',peptide,"S");
end
end
if seq(3*i-1)=="G"
if seq(3*i)=="A"
flag=1;
break;
elseif seq(3*i)=="T"
peptide=sprintf('%s%s',peptide,"C");
elseif seq(3*i)=="C"
peptide=sprintf('%s%s',peptide,"C");
elseif seq(3*i)=="G"
peptide=sprintf('%s%s',peptide,"W");
end
end
end
if seq(3*i-2)=="C"
if seq(3*i-1)=="A"
if seq(3*i)=="A"
peptide=sprintf('%s%s',peptide,"Q");
elseif seq(3*i)=="T"
peptide=sprintf('%s%s',peptide,"H");
elseif seq(3*i)=="C"
peptide=sprintf('%s%s',peptide,"H");
elseif seq(3*i)=="G"
peptide=sprintf('%s%s',peptide,"Q");
end
end
if seq(3*i-1)=="T"
if seq(3*i)=="A"
peptide=sprintf('%s%s',peptide,"L");
elseif seq(3*i)=="T"
peptide=sprintf('%s%s',peptide,"L");
elseif seq(3*i)=="C"
peptide=sprintf('%s%s',peptide,"L");
elseif seq(3*i)=="G"
peptide=sprintf('%s%s',peptide,"L");
end
end
if seq(3*i-1)=="C"
if seq(3*i)=="A"
peptide=sprintf('%s%s',peptide,"P");
elseif seq(3*i)=="T"
peptide=sprintf('%s%s',peptide,"P");
elseif seq(3*i)=="C"
peptide=sprintf('%s%s',peptide,"P");
elseif seq(3*i)=="G"
peptide=sprintf('%s%s',peptide,"P");
end
end
if seq(3*i-1)=="G"
if seq(3*i)=="A"
peptide=sprintf('%s%s',peptide,"R");
elseif seq(3*i)=="T"
peptide=sprintf('%s%s',peptide,"R");
elseif seq(3*i)=="C"
peptide=sprintf('%s%s',peptide,"R");
elseif seq(3*i)=="G"
peptide=sprintf('%s%s',peptide,"R");
end
end
end
if seq(3*i-2)=="G"
if seq(3*i-1)=="A"
if seq(3*i)=="A"
peptide=sprintf('%s%s',peptide,"E");
elseif seq(3*i)=="T"
peptide=sprintf('%s%s',peptide,"D");
elseif seq(3*i)=="C"
peptide=sprintf('%s%s',peptide,"D");
elseif seq(3*i)=="G"
peptide=sprintf('%s%s',peptide,"E");
end
end
if seq(3*i-1)=="T"
if seq(3*i)=="A"
peptide=sprintf('%s%s',peptide,"V");
elseif seq(3*i)=="T"
peptide=sprintf('%s%s',peptide,"V");
elseif seq(3*i)=="C"
peptide=sprintf('%s%s',peptide,"V");
elseif seq(3*i)=="G"
peptide=sprintf('%s%s',peptide,"V");
end
end
if seq(3*i-1)=="C"
if seq(3*i)=="A"
peptide=sprintf('%s%s',peptide,"A");
elseif seq(3*i)=="T"
peptide=sprintf('%s%s',peptide,"A");
elseif seq(3*i)=="C"
peptide=sprintf('%s%s',peptide,"A");
elseif seq(3*i)=="G"
peptide=sprintf('%s%s',peptide,"A");
end
end
if seq(3*i-1)=="G"
if seq(3*i)=="A"
peptide=sprintf('%s%s',peptide,"G");
elseif seq(3*i)=="T"
peptide=sprintf('%s%s',peptide,"G");
elseif seq(3*i)=="C"
peptide=sprintf('%s%s',peptide,"G");
elseif seq(3*i)=="G"
peptide=sprintf('%s%s',peptide,"G");
end
end
end
if flag==1;
fprintf("Be careful,met the terminator when the Ordinal is %d",i)
break;
end
end
peptide=strrep(peptide," ","");%去除空白字符串数组生成时的空格
fprintf("The second type for sequence of peptide is %s\n",peptide)%输出肽链氨基酸信息
%%%%第三种解释方式
long_peptide = (base_number-2-mod(base_number-2,3))/3; %计算肽链长度,为定义空白字符串数组做准备
fprintf("#3.The length of peptide ignore Terminator is %d\n",long_peptide)%输出肽链长度信息
peptide=" ";%生成空白字符串数组,以便将对应的字符附加于其上
for i=1:base_number-2%通过去掉基因序列的前两个字符模拟密码子不读取前两个碱基的情况
seq(i)=seq(i+1);
end
seq(i+1)="";%去掉字符串数组中多余的最后一个字符
for i=1:long_peptide %翻译操作
flag=0; %与后面的"flag=1"对应,实现终止子功能
if seq(3*i-2)=="A"
if seq(3*i-1)=="A"
if seq(3*i)=="A"
peptide=sprintf('%s%s',peptide,"K");
elseif seq(3*i)=="T"
peptide=sprintf('%s%s',peptide,"N");
elseif seq(3*i)=="C"
peptide=sprintf('%s%s',peptide,"N");
elseif seq(3*i)=="G"
peptide=sprintf('%s%s',peptide,"K");
end
end
if seq(3*i-1)=="T"
if seq(3*i)=="A"
peptide=sprintf('%s%s',peptide,"I");
elseif seq(3*i)=="T"
peptide=sprintf('%s%s',peptide,"I");
elseif seq(3*i)=="C"
peptide=sprintf('%s%s',peptide,"I");
elseif seq(3*i)=="G"
peptide=sprintf('%s%s',peptide,"M");
end
end
if seq(3*i-1)=="C"
if seq(3*i)=="A"
peptide=sprintf('%s%s',peptide,"T");
elseif seq(3*i)=="T"
peptide=sprintf('%s%s',peptide,"T");
elseif seq(3*i)=="C"
peptide=sprintf('%s%s',peptide,"T");
elseif seq(3*i)=="G"
peptide=sprintf('%s%s',peptide,"T");
end
end
if seq(3*i-1)=="G"
if seq(3*i)=="A"
peptide=sprintf('%s%s',peptide,"R");
elseif seq(3*i)=="T"
peptide=sprintf('%s%s',peptide,"S");
elseif seq(3*i)=="C"
peptide=sprintf('%s%s',peptide,"S");
elseif seq(3*i)=="G"
peptide=sprintf('%s%s',peptide,"R");
end
end
end
if seq(3*i-2)=="T"
if seq(3*i-1)=="A"
if seq(3*i)=="A"
flag=1;
break;
elseif seq(3*i)=="T"
peptide=sprintf('%s%s',peptide,"Y");
elseif seq(3*i)=="C"
peptide=sprintf('%s%s',peptide,"Y");
elseif seq(3*i)=="G"
flag=1;
break;
end
end
if seq(3*i-1)=="T"
if seq(3*i)=="A"
peptide=sprintf('%s%s',peptide,"L");
elseif seq(3*i)=="T"
peptide=sprintf('%s%s',peptide,"F");
elseif seq(3*i)=="C"
peptide=sprintf('%s%s',peptide,"F");
elseif seq(3*i)=="G"
peptide=sprintf('%s%s',peptide,"L");
end
end
if seq(3*i-1)=="C"
if seq(3*i)=="A"
peptide=sprintf('%s%s',peptide,"S");
elseif seq(3*i)=="T"
peptide=sprintf('%s%s',peptide,"S");
elseif seq(3*i)=="C"
peptide=sprintf('%s%s',peptide,"S");
elseif seq(3*i)=="G"
peptide=sprintf('%s%s',peptide,"S");
end
end
if seq(3*i-1)=="G"
if seq(3*i)=="A"
flag=1;
break;
elseif seq(3*i)=="T"
peptide=sprintf('%s%s',peptide,"C");
elseif seq(3*i)=="C"
peptide=sprintf('%s%s',peptide,"C");
elseif seq(3*i)=="G"
peptide=sprintf('%s%s',peptide,"W");
end
end
end
if seq(3*i-2)=="C"
if seq(3*i-1)=="A"
if seq(3*i)=="A"
peptide=sprintf('%s%s',peptide,"Q");
elseif seq(3*i)=="T"
peptide=sprintf('%s%s',peptide,"H");
elseif seq(3*i)=="C"
peptide=sprintf('%s%s',peptide,"H");
elseif seq(3*i)=="G"
peptide=sprintf('%s%s',peptide,"Q");
end
end
if seq(3*i-1)=="T"
if seq(3*i)=="A"
peptide=sprintf('%s%s',peptide,"L");
elseif seq(3*i)=="T"
peptide=sprintf('%s%s',peptide,"L");
elseif seq(3*i)=="C"
peptide=sprintf('%s%s',peptide,"L");
elseif seq(3*i)=="G"
peptide=sprintf('%s%s',peptide,"L");
end
end
if seq(3*i-1)=="C"
if seq(3*i)=="A"
peptide=sprintf('%s%s',peptide,"P");
elseif seq(3*i)=="T"
peptide=sprintf('%s%s',peptide,"P");
elseif seq(3*i)=="C"
peptide=sprintf('%s%s',peptide,"P");
elseif seq(3*i)=="G"
peptide=sprintf('%s%s',peptide,"P");
end
end
if seq(3*i-1)=="G"
if seq(3*i)=="A"
peptide=sprintf('%s%s',peptide,"R");
elseif seq(3*i)=="T"
peptide=sprintf('%s%s',peptide,"R");
elseif seq(3*i)=="C"
peptide=sprintf('%s%s',peptide,"R");
elseif seq(3*i)=="G"
peptide=sprintf('%s%s',peptide,"R");
end
end
end
if seq(3*i-2)=="G"
if seq(3*i-1)=="A"
if seq(3*i)=="A"
peptide=sprintf('%s%s',peptide,"E");
elseif seq(3*i)=="T"
peptide=sprintf('%s%s',peptide,"D");
elseif seq(3*i)=="C"
peptide=sprintf('%s%s',peptide,"D");
elseif seq(3*i)=="G"
peptide=sprintf('%s%s',peptide,"E");
end
end
if seq(3*i-1)=="T"
if seq(3*i)=="A"
peptide=sprintf('%s%s',peptide,"V");
elseif seq(3*i)=="T"
peptide=sprintf('%s%s',peptide,"V");
elseif seq(3*i)=="C"
peptide=sprintf('%s%s',peptide,"V");
elseif seq(3*i)=="G"
peptide=sprintf('%s%s',peptide,"V");
end
end
if seq(3*i-1)=="C"
if seq(3*i)=="A"
peptide=sprintf('%s%s',peptide,"A");
elseif seq(3*i)=="T"
peptide=sprintf('%s%s',peptide,"A");
elseif seq(3*i)=="C"
peptide=sprintf('%s%s',peptide,"A");
elseif seq(3*i)=="G"
peptide=sprintf('%s%s',peptide,"A");
end
end
if seq(3*i-1)=="G"
if seq(3*i)=="A"
peptide=sprintf('%s%s',peptide,"G");
elseif seq(3*i)=="T"
peptide=sprintf('%s%s',peptide,"G");
elseif seq(3*i)=="C"
peptide=sprintf('%s%s',peptide,"G");
elseif seq(3*i)=="G"
peptide=sprintf('%s%s',peptide,"G");
end
end
end
if flag==1;
fprintf("Be careful,met the terminator when the Ordinal is %d",i)
break;
end
end
peptide=strrep(peptide," ","");%去除空白字符串数组生成时的空格
fprintf("The third type for sequence of peptide is %s\n",peptide)%输出肽链氨基酸信息© 版权声明
文章版权归作者所有,未经允许请勿转载。
THE END


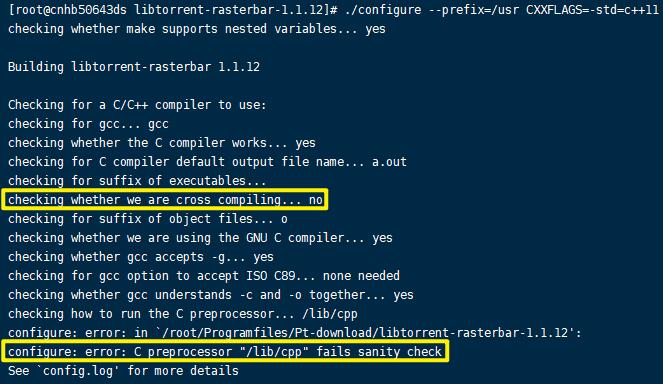



暂无评论内容